

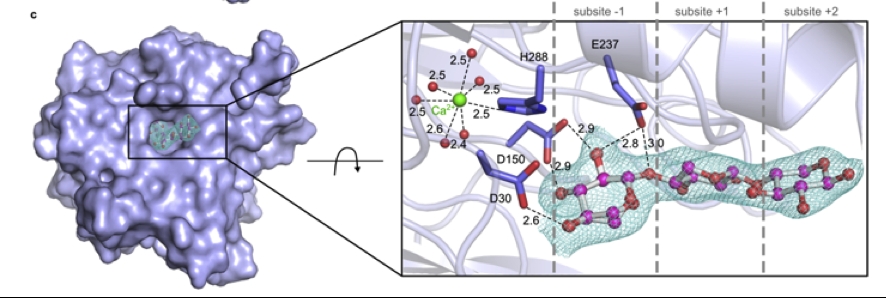
Estrutura cristalográfica revela enzima momentos antes de sua atuação sobre o substrato; detalhe na enzima (em roxo) se refere ao sítio ativo, exatamente onde o substrato se liga (imagem: LNBR-CNPEM)
Publicado em 15/03/2021
Agência FAPESP* – No jargão científico, é denominado “itinerário catalítico” o conjunto de modificações químicas e estruturais que um determinado substrato sofre pela ação de uma enzima até ser clivado (quebrado em pedaços) e um novo produto ser formado. Até pouco tempo, considerava-se que o itinerário catalítico era único para cada par de enzima-substrato. Mas esse paradigma acaba de ser quebrado por pesquisadores do Laboratório Nacional de Biorrenováveis (LNBR), que integra o Centro Nacional de Pesquisa em Energia e Materiais (CNPEM), em Campinas.
Os resultados da pesquisa, apoiada pela FAPESP, foram divulgados na revista Nature Communications.
Por meio de cálculos quânticos e experimentos com super-resolução, os cientistas mostraram que algumas enzimas de grande importância industrial – como as hidrolases glicosídicas – podem funcionar por itinerários catalíticos alternativos e viáveis do ponto de vista termodinâmico.
O trabalho revela que essas enzimas que atuam sobre a hemicelulose (polissacarídeo presente na parede celular das plantas) – e que são essenciais, portanto, para o processamento de resíduos agroindustriais – são capazes de clivar ligações glicosídicas por duas rotas catalíticas, algo inesperado em biocatálise de carboidratos. Essa descoberta abre novos horizontes a respeito do funcionamento molecular de uma classe de enzimas de grande relevância industrial e traz impactos imediatos no desenvolvimento teórico da área de biocatálise.
“Esses resultados, além de modificar nosso entendimento no campo teórico da biocatálise de carboidratos, elevam nossa capacidade de racionalmente ‘engenheirar’ enzimas visando aplicações biotecnológicas”, esclarece Mario Murakami, diretor científico do LNBR-CNPEM e coordenador do projeto.
Na prática, o estudo permitirá que outros pesquisadores encontrem formas para redesenhar o sítio ativo (região da enzima responsável por ligar e converter o substrato em um produto), bem como para criar moléculas análogas ao substrato capazes de inibir ações de determinadas enzimas.
Isso pode ter aplicações que vão da produção industrial de químicos biorrenováveis (obtidos a partir de plantas) à área da saúde.
“Pensemos em um profissional trabalhando com uma célula. O pesquisador identifica uma atividade enzimática que precisa ser inibida. A partir do conhecimento obtido com o desenho racional, é possível criar este inibidor customizado”, explica Mariana Morais, pesquisadora do LNBR-CNPEM e autora principal do artigo.
No estudo, os cientistas adotaram uma técnica conhecida como cristalografia de raios X – que consiste em cristalizar proteínas e observar como esse cristal difrata a radiação emitida sobre ele. Para isso, usaram o estado da arte em instrumentação disponível no Laboratório Nacional de Luz Síncrotron (LNLS-CNPEM). Já as simulações computacionais baseadas em cálculos quânticos foram realizadas na rede espanhola de supercomputadores e na Universidade de Barcelona. Os demais experimentos foram feitos nas instalações do LNBR e do Laboratório Nacional de Biociências (LNBio). A investigação contou com apoio da FAPESP (15/26982-0, 16/19995-0 e 18/22138-8), do Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq) e de agências financiadoras da Europa.
O artigo Two distinct catalytic pathways for GH43 xylanolytic enzymes unveiled by X-ray and QM/MM simulations pode ser lido em www.nature.com/articles/s41467-020-20620-3.
* Com informações da Assessoria de Comunicação do CNPEM